人体内每个细胞都包含相同的基因序列,但每个细胞仅表达这些基因的特定部分。这种细胞特异性基因表达模式确保了脑细胞与皮肤细胞的不同特性,主要由染色质的三维结构决定。遗传物质控制着每个基因的可及性。近年来,高通量测序和成像技术的发展显著提高了研究单个细胞基因组组织的能力。然而,现有方法通常费力且耗时,阻碍了对不同细胞环境中染色质构象变异性的全面分析。
麻省理工学院的化学家们开发了一种使用生成式人工智能的新方法,可以快速预测这些 3D 基因组结构。该技术可以在几分钟内预测数千个结构,比现有的实验方法快得多。他们的研究题为「ChromoGen: Diffusion model predicts single-cell chromatin conformations」,发表在2025年1月31日的《Science Advances》上。
麻省理工学院化学副教授 Bin Zhang 表示:“我们的目标是尝试从潜在的 DNA 序列中预测三维基因组结构。现在我们可以做到这一点,这使得它能够与尖端的实验技术相媲美,并开辟了许多有趣的研究机会。”
了解基因组的三维(3D)组织对于揭示其功能复杂性和在基因调控中的作用至关重要。分子生物学技术的进步为基因组构象提供了宝贵的见解,揭示了染色质结构如何促进基因周围空间环境的建立,有助于在适当的时间募集适当的分子来协调转录。最近,深度学习技术为直接从测序数据预测单细胞染色质构象提供了有潜力的研究方向。
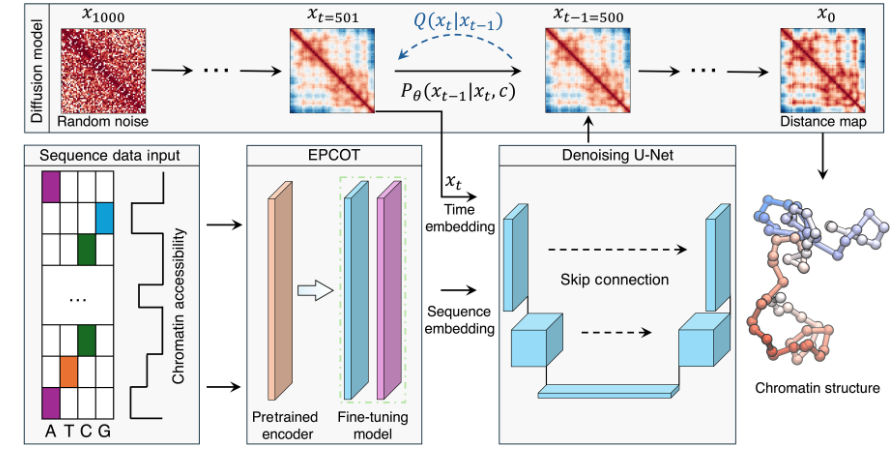
为了预测单个染色质结构,团队引入了 ChromoGen,这是一种扩散模型,已被证明在文本到图像应用以及预测配体和蛋白质分子的 3D 坐标方面具有很强的能力。微调的 EPCOT 模型将 DNA 序列和 DNase-seq 数据转换为低维数字嵌入,然后使用无分类器指导的去噪扩散概率模型(DDPM)对采样的分布进行条件处理。此外,距离图自然不受 3D 构象的旋转和平移的影响,许多神经网络架构难以理解,并且距离图可以轻松转换为笛卡尔坐标。
为了验证 ChromoGen 的性能,研究人员通过组合来自更广泛数据集中每个区域的 200 个生成的结构来近似一个通用集成,并计算了由各种序列长度分隔的基因座之间空间距离的概率分布。结果显示,ChromoGen 能够准确捕获在单细胞染色质结构中观察到的构象分布。生成的构象涵盖了广泛的构象和结构基序,验证了其高度复杂结构的能力。
为了定量评估生成结构和实验结构之间的一致性,团队对预测数据集所包含的整个基因组区域进行了多次分析。结果显示,使用生成的和 Dip-C 构象计算的中位距离图始终具有强相关性。此外,ChromoGen 还能准确预测来自其训练所针对的细胞类型以外的细胞类型的数据,表明它可能有助于分析不同细胞类型的染色质结构差异及其对功能的影响。
ChromoGen 是一种智能框架,能够有效生成区域和细胞类型特异性的染色质构象。经过训练后,该模型可以在比 Hi-C 或其他实验技术更快的时间尺度上生成预测。例如,在一个 GPU 上,它可以20分钟内生成特定区域的一千个结构。研究人员表示,他们希望通过这个模型解决许多有趣的问题,例如探索特定 DNA 序列中的突变如何改变染色质构象。
本文来源: 机器之心【阅读原文】
机器之心【阅读原文】